Bofvirusgenotypering is gebaseerd op de genetische variabiliteit van het small hydrophobic (SH)-gen van het virus. De meest recente bofuitbraken in Nederland blijken vrijwel steeds te worden veroorzaakt door bofvirusgenotype G. De geringe variatie in het SH (small hydrophobic)-gen binnen bof-virusgenotype G maakt dit gen echter ongeschikt om endemische transmissie te onderscheiden van geïmporteerde bofvirussen. Daarom hebben we naast het SH-gen ook het fusie(F)gen en het hemagglutinine-neuraminidase(HN)gen geanalyseerd van Nederlandse bofvirussen uit 2015 en 2016. Op deze wijze kunnen we aantonen dat cases met een identieke sequentie tot hetzelfde uitbraakcluster behoren, ook bij afwezigheid van een epidemiologische link.
content
Auteurs: S. Gouma, I. Veldhuijzen, R. van Binnendijk
Infectieziekten Bulletin: december 2016, jaargang 27, nummer 10
Bofuitbraken
De invoering van het vaccin tegen bof, mazelen en rodehond (BMR) in het Rijksvaccinatieprogramma (RVP) in 1987 heeft de ziektelast van bof sterk teruggedrongen. In het afgelopen decennium zijn er wereldwijd echter verscheidene bofuitbraken onder BMR (bof, mazelen,rodehond)-gevaccineerde personen geweest, waaronder ook in Nederland. De meeste bofpatiënten tijdens deze uitbraken waren jongvolwassenen. De eerste bofuitbraak in Nederland onder BMR-gevaccineerde personen was in 2004 op de Hotelschool in Den Haag. (1) Na deze uitbraak was er een cluster in Brabant, dat net als het cluster in Den Haag door bofvirusgenotype G werd veroorzaakt. In 2007-2009 was er een genotype D-bofuitbraak onder bevindelijk gereformeerde groeperingen met een lage vaccinatiegraad. (2) De grootste genotype G-bofuitbraak in Nederland begon in 2009 en breidde zich snel uit na een studentenfeest in februari 2010, resulterend in een landelijke uitbraak die tot 2012 aanhield. (3,4) Sinds 2012 zijn er alleen nog enkele lokale bofuitbraken geweest.
Bofsurveillance door genotypering
Genotypering van de circulerende bofvirussen speelt een belangrijke rol in bofsurveillance. Zo kan een aan vaccingerelateerde bof (genotype A) worden vastgesteld en kunnen geografische verschuivingen van de verschillende genotypen door de tijd heen worden gedetecteerd. (5) Op basis van het SH (small hydrophobic)-gen en het HN (hemagglutinine-neuraminidase)-gen zijn 12 genotypen gedefinieerd. (6) Het SH-gen is relatief klein en wordt meestal gebruikt voor typering vanwege de grote variabiliteit in dit gen. (7) Binnen genotype G-stammen blijkt de variatie in het SH-gen echter gering te zijn, wat resulteerde in grote clusters met identieke SH-gen-sequenties tijdens de recente genotype G-bofuitbraken in Nederland. (3)
Om de moleculaire resolutie te vergroten hebben wij naast het SH-gen, dat codeert voor een membraaneiwit, ook de nucleotidesequenties van het F-gen en HN-gen geanalyseerd. Het F-gen en het HN-gen zijn groter dan het SH-gen en coderen voor de oppervlakte-eiwitten van het virus, die een belangrijke rol spelen in de pathogenese en immuniteit. Door de sequenties van deze 3 genen te combineren wordt een grotere genetische diversiteit zichtbaar en daarmee kan worden vastgesteld of bepaalde gevallen behoren tot een specifiek uitbraakcluster, of dat er sprake is van een andere bron van infectie. Op deze manier kan worden onderzocht of de bofgevallen in 2015 en 2016 losstaande gevallen zijn of dat er sprake is van circulatie van het virus over een langere periode.
De gegevens over bofpatiënten die gemeld werden in Osiris tussen 1 januari 2015 en 30 juni 2016 zijn meegenomen in de analyse als de eerste ziektedag bekend was. Indien de eerste ziektedag niet vermeld was in Osiris is deze bij benadering achterhaald op basis van de diagnostische aanvragen. Zowel klinische materialen ingezonden naar het RIVM voor bofdiagnostiek die positief zijn getest op bof RNA (ribonucleic acid) (ribonucleïnezuur) met behulp van PCR (polymerase chain reaction) (polymerase chain reaction), als ook materialen die elders positief zijn getest en zijn ingezonden voor verdere moleculaire analyse, zijn gesequenced (Tabel 1). Het sequencen van het F-gen, SH-gen en HN-gen werd uitgevoerd zoals eerder is gepubliceerd. (8)
In 2015 en de eerste helft van 2016 werden 131 bofpatiënten in Osiris gerapporteerd (Tabel 1). Van 128 patiënten was de eerste ziektedag via Osiris gerapporteerd en van 3 andere patiënten kon de eerste ziektedag bij benadering worden achterhaald op basis van de diagnostische aanvragen.
Boftransmissie in 2015
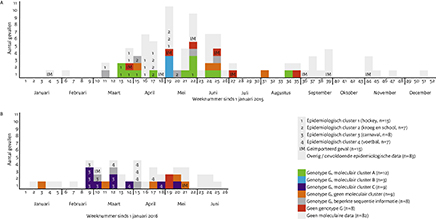
In 2015 werden er 2 epidemiologische bofclusters waargenomen. Cluster 1 (n=13) was gerelateerd aan een hockeyvereniging in Zuid-Holland. Tot dit cluster behoorden onder andere enkele teamgenoten van de indexpatiënt. De indexpatiënt uit dit cluster kreeg op medio maart de eerste klachten en de laatste patiënt uit dit cluster kreeg eind mei bofklachten.
Cluster 2 (n=7) was oorspronkelijk gerelateerd aan een café in Zuid-Holland en de indexpatiënt uit dit cluster kreeg begin april klachten. Vervolgens breidde dit cluster zich uit naar een MBO (middelbaar beroepsonderwijs) (Middelbaar Beroepsonderwijs)-college in Zuid-Holland omdat 1 van de bofpatiënten hier studeerde (Figuur 1A). De laatste patiënt uit dit cluster kreeg medio mei bofklachten.
Naast deze 2 epidemiologische bofclusters zijn er 11 bofpatiënten gerapporteerd, waarbij besmetting met het bofvirus óf in het buitenland had plaatsgevonden (primair) óf via een ander persoon die in het buitenland geïnfecteerd was geraakt (secundair).
Van de 30 patiënten in 2015 waarvan het bofvirus kon worden getypeerd, waren 24 patiënten geïnfecteerd met een bofvirusgenotype G. Op basis van de nucleotidesequenties waren 2 clusters te onderscheiden: cluster A (n=12) en cluster B (n=3).
Patiënten behorend tot cluster A werden ziek tussen eind maart en half juni, met nog een enkele geïdentificeerde patiënt in augustus. Cluster A bestond uit 5 patiënten uit epidemiologisch cluster 1. Van de overige patiënten uit cluster 1 waren geen moleculaire data beschikbaar. Ook bleek een patiënt uit moleculair cluster A met een eerste ziektedag eind april tot epidemiologisch cluster 2 te behoren, wat aantoont dat de epidemiologische clusters 1 en 2 aan elkaar gelinkt waren (Figuur 1A). Van 2 andere patiënten uit epidemiologisch cluster 2 kon alleen het SH-gen worden getypeerd, waardoor het niet mogelijk was om deze patiënten verder in een moleculair cluster in te delen. Van de overige patiënten uit cluster 2 waren geen moleculaire data beschikbaar.
Moleculair cluster B bestond uit 3 patiënten die klachten kregen op 7-9 mei, maar niet geografisch en epidemiologisch gelinkt waren. Gezien de incubatietijd van 2-4 weken is het echter aannemelijk dat dit moleculaire cluster in werkelijkheid meer patiënten bevatte. Omdat de SH-gen- sequentie in monsters behorende tot cluster B identiek was aan de SH-sequentie in monsters uit cluster A, kon alleen op basis van het F-gen en het HN-gen onderscheid tussen deze moleculaire clusters worden gemaakt.
Tabel 1 Gerapporteerde bofpatiënten in Osiris (n=131) 2015 (download de pdf voor een vergrote weergave).
Figuur 1 Overzicht van de bofpatiënten gerapporteerd in Osiris. (A) Overzicht van de 88 bofpatiënten in 2015. (B) Overzicht van de 43 bofpatiënten in de eerste 6 maanden van 2016 (download de pdf voor een vergrote weergave).
Boftransmissie in 2016
In de eerste 6 maanden van 2016 werden opnieuw 2 epidemiologische bofclusters geïdentificeerd, beide in Noord-Brabant: cluster 3 (n=8) was gerelateerd aan carnaval en cluster 4 (n=7) was gerelateerd aan een voetbalvereniging. De eerste patiënt uit cluster 3 kreeg eind februari bofklachten en de laatste patiënt uit dit cluster kreeg half maart klachten. Patiënten uit cluster 4 kregen klachten tussen half maart en begin mei (Figuur 1B). Naast deze 2 epidemiologische bofclusters werden 2 patiënten gerapporteerd die voordat ze ziek werden in het buitenland waren geweest en daar contact hadden gehad met een bofpatiënt.
Van de 19 patiënten in 2016 waarvan het bofvirus getypeerd kon worden, waren 17 patiënten geïnfecteerd met een bofvirusgenotype G. De overige 2 patiënten kregen bofklachten op respectievelijk 2 en 5 mei door een ander bofvirusgenotype C, maar wel beide met een identieke sequentie. Het ligt daarom voor de hand dat deze patiënten, die uit dezelfde regio komen, via een gemeenschappelijke bron besmet waren geraakt. Van de 17 patiënten die geïnfecteerd waren met een bofvirusgenotype G behoorden 9 patiënten tot moleculair cluster C. Zes patiënten uit dit cluster behoorden tot epidemiologisch cluster 3 en 2 patiënten behoorden tot epidemiologisch cluster 4, wat opnieuw aantoont dat deze 2 epidemiologische clusters gelinkt waren (Figuur 1B).
Analyse van de gecombineerde nucleotidesequenties van het F-gen, SH-gen en HN-gen vergroot de moleculaire resolutie en maakt het mogelijk om endemische circulatie te onderscheiden van individuele patiënten met bof. Zo konden tijdens de bofuitbraak in 2015 op basis van de aanvullende F-gen- en HN-gensequenties 2 moleculaire bofclusters worden onderscheiden, terwijl de SH-gensequenties in deze clusters identiek waren. Bofpatiënten die tot een epidemiologisch cluster behoren hebben identieke F-gen-, SH-gen- en HN-gensequenties, waardoor het waarschijnlijk is dat patiënten die geïnfecteerd zijn met een bofvirus met een afwijkende sequentie, niet tot het cluster behoren. Moleculair cluster A was bijvoorbeeld gedurende een langere tijd (maart t/m juni 2015) consistent aantoonbaar en dit is een sterke aanwijzing voor endemische transmissie van dit type bofvirus in deze periode. In augustus 2015 werd nog een enkele patiënt getraceerd met deze sequentie. Dit zou kunnen betekenen dat er veel gevallen zijn gemist en dat dit virus veel langer endemisch is geweest in Nederland, of dat er sprake is van een nieuwe bron van infectie met dit type virus.
Het feit dat er in 2015 meerdere patiënten waren geïnfecteerd met een ander genotype duidt erop dat er meerdere bronnen van infectie zijn. Een deel van deze besmettingen kon op basis van de epidemiologische gegevens worden geoormerkt als geïmporteerd dan wel importgerelateerd. Verder kon bij een aanzienlijk deel van de gediagnosticeerde patiënten het bofvirusgenotype niet worden getypeerd omdat er geen adequate klinische materialen voorhanden waren.
In 2016 werd opnieuw een uniek moleculair cluster (cluster C) geïdentificeerd. Hiertoe behoorden alle patiënten uit de epidemiologische clusters 3 en 4, waarvan moleculaire
data beschikbaar waren. Ook deze sequentie werd gedurende langere tijd (februari t/m mei 2016) consistent aangetoond en vormt zo een sterke aanwijzing voor endemische transmissie van dit type bofvirus in deze periode.
Ook in 2016 werden meerdere patiënten geïnfecteerd met een afwijkend bofvirusgenotype of een bofvirusgenotype G met een afwijkende sequentie, wat duidt op meerdere introducties van het virus.
Omdat de beschikbare moleculaire data van circulerende bofvirussen wereldwijd beperkt zijn, kunnen we met deze eerste analyse nog niet volledig uitsluiten dat introductie met een bofvirusstam met dezelfde sequentie meerdere malen heeft plaatsgevonden. Deze uitgebreide typeringstool maakt het echter wel mogelijk om endemische transmissie van bof vast te stellen, indien de typering op regelmatige basis wordt uitgevoerd. Onze typering laat zien dat er frequent infecties plaatsvinden met bofvirus vanuit verschillende bronnen, die soms leiden tot kleine lokale bofuitbraken die meerdere maanden kunnen aanhouden. Op deze manier wordt het mogelijk om grotere bofuitbraken tijdig te signaleren en om het effect van eventuele maatregelen op virustransmissie tijdens een uitbraak te meten.
Auteurs
S. Gouma1,2, I. Veldhuijzen1, R. van Binnendijk1
- Centrum Infectieziektebestrijding, RIVM, Bilthoven
- Afdeling Viroscience, Erasmus MC (Erasmus University Medical Center), Rotterdam
Correspondentie:
- Brockhoff HJ, Mollema L, Sonder GJB, Postema CA, van Binnendijk RS (respiratoir syncytieel), Kohl RHG, et al. Mumps outbreak in a highly vaccinated student population, The Netherlands, 2004. Vaccine. 2010;28(17):2932–6.
- Wielders CC, van Binnendijk RS, Snijders BE, Tipples GA, Cremer J, Fanoy E, et al. Mumps epidemic in orthodox religious low-vaccination communities in the Netherlands and Canada, 2007 to 2009. Eurosurveillance. 2011;16(41):19989.
- Gouma S, Sane J, Gijselaar D, Cremer J, Hahné S, Koopmans M, et al. Two major mumps genotype G variants dominated recent mumps outbreaks in the Netherlands (2009-2012). J Gen Virol. 2014;95(Pt 5):1074–82.
- Sane J, Gouma S, Koopmans M, Melker H De, Swaan C, Binnendijk R Van, et al. Epidemic of mumps among vaccinated persons, the Netherlands, 2009-2012. Emerg Infect Dis. 2014;20(4):643–8.
- Jin L, Brown DWG, Litton PA, White JM (Joint meeting). Genetic diversity of mumps virus in oral fluid specimens: application to mumps epidemiological study. J Infect Dis. 2004;189(6):1001–8.
- World Health Organization. Mumps virus nomenclature update: 2012. Wkly Epidemiol Rec. 2012;87:217–24.
- Jin L, Orvell C, Myers R, Rota P, Nakayama T, Forcic D, et al. Genomic diversity of mumps virus and global distribution of the 12 genotypes. Rev Med Virol. 2015;25(2):85–101.
- Gouma S, Cremer J, Parkkali S, Veldhuijzen I, van Binnendijk RS, Koopmans MPG (mpg). Mumps virus F gene and HN (hemagglutinine-neuraminidase) gene sequencing as a molecular tool to study mumps virus transmission. Ingediend.